이번 강의의 주제는 '건강검진 데이터로 가설검정하기'이며 해당 데이터를 다운받기 위해 아래 공공데이터 포털에서 접속한다. 그리고 하단의 주기성 과거 데이터 (20건)의 더보기 버튼을 클릭한 후 건강검진정보(2017) csv 파일을 다운받는다.
https://www.data.go.kr/data/15007122/fileData.do

전체 결과 코드 : https://github.com/vavana619/health_analysis_input
GitHub - vavana619/health_analysis_input
Contribute to vavana619/health_analysis_input development by creating an account on GitHub.
github.com
1. 음주 여부에 따라 건강검진 수치 차이가 있을까? (가설 1)
2. 신장과 허리둘레의 크기는 체중과 상관관계가 있을까? (가설 2)
→ 분석을 통해 가설을 검정하기
3. 라이브러리 로드
import pandas as pd # 분석
import numpy as np # 수치계산
import seaborn as sns # 시각화
import matplotlib.pyplot as plt
# 구 버전의 주피터 노트북에서는 %matplotlib inline 설정이 되어야 노트북 안에서 그래프를 시각화함
%matplotlib inline
4. 한글폰트 설정
한글폰트를 설정해 주지 않으면 그래프 상에서 한글이 깨져보인다. → 한글이 출력될 수 있도록 폰트 설정하기
import os
if os.name == "posix" : # Mac 사용자라면
plt.rc("font", family="AppleGothic")
else:
plt.rc("font", family="Malgun Gothic")
# 마이너스 폰트 깨지는 문제에 대한 대처
plt.rc("axes", unicode_minus=False)레티나(retina) 설정을 해주면 글씨가 좀 더 선명하게 보인다. 그리고 폰트의 주변이 흐릿하게 보이는 것을 방지해주는 효과가 있다.
%config InlineBackend.figure_format = "retina"
5. 데이터 불러오기
cp949 : EUC-KR의 확장이며 EUC-KR에서 표현할 수 없는 한글도 표현할 수 있음
df = pd.read_csv("C:/data/NHIS_OPEN_GJ_2017.csv", encoding="cp949")
df.shape(1000000, 34)
1) 데이터 미리보기
df.head()
df.tail()
df.sample() # 한 개만 랜덤하게 가져옴
6. 기본정보 보기
info() 함수를 사용하여 데이터 결측치를 살펴본다.
df.info()

columns을 이용하여 컬럼만 따로 모아서 출력한다.
df.columns
dtypes을 이용하여 데이터 형식만 따로 모아서 출력한다.
df.dtypes
7. 결측치 보기
- isnull() 함수를 통해 결측치를 boolean 값(True/False) 값으로 표시
- isna() 함수로도 결측치 여부를 확인할 수 있음
- sum() 함수를 통해 column마다의 결측치 수를 계산하여 표시
# 결과 동일
df.isnull().sum()
df.isna().sum()
pandas에 내장된 plot을 통해 시각화하기
df.isnull().sum().plot.barh(figsize=(10, 9))
8. 일부 데이터 요약하기
- 여러 컬럼 가져오기 : 리스트 형태 [ ] 로 감싸주기 (데이터프레임 형태)
- "(혈청지오티)ALT", (혈청지오티)AST" 를 가져와 미리보기
df[["(혈청지오티)ALT", "(혈청지오티)AST"]].head()
"(혈청지오티)ALT", (혈청지오티)AST" 를 요약하기
df[["(혈청지오티)ALT", "(혈청지오티)AST"]].describe()
9. value_counts로 값 집계하기
"성별코드"를 그룹화하고 개수를 집계하기 - 1: 남성 / 2 : 여성
df["성별코드"].value_counts()
"흡연상태"로 그룹화하고 개수를 집계 - 1: 흡연 / 3: 금연 / 2: 흡연했다가 금연
df["흡연상태"].value_counts()
10. groupby와 pivot_table 사용하기
1) groupby
"성별코드"로 그룹화 한 데이터를 세어보기 ("가입자일련번호"만 출력하도록 설정) -> count()
df.groupby(["성별코드"])["가입자일련번호"].count()
"성별코드"와 "음주여부"로 그룹화를 하고 갯수를 세어보기 ("가입자일련번호"만 출력하도록 설정)
df.groupby(["성별코드", "음주여부"])["가입자일련번호"].count()
"성별코드"와 "음주여부"로 그룹화를 하고 감마지티피의 평균을 구하기 -> mean()
df.groupby(["성별코드", "음주여부"])["감마지티피"].mean()
"성별코드"와 "음주여부"로 그룹화를 하고 감마지티피의 요약수치 구하기 -> describe()
df.groupby(["성별코드", "음주여부"])["감마지티피"].describe()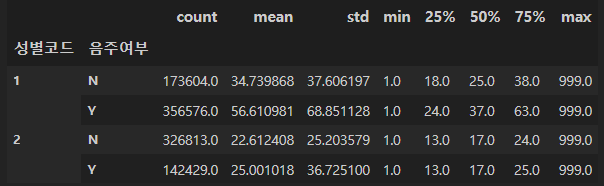
agg를 사용하면 여러 수치를 함께 구할 수 있음
df.groupby(["성별코드", "음주여부"])["감마지티피"].agg(["count", "mean", "median"])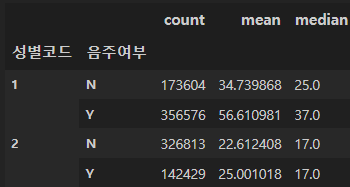
2) pivot_table
데이터프레임명.pivot_table(index="행", values="열", aggfuc="함수명")
- "음주여부"에 따른 그룹화된 수를 피벗테이블로 구하기
- groupby와 결과는 같지만 groupby는 시리즈 형태로 출력됨 -> 속도가 더 빠름
- 그러나 직관적으로 pivot_table이 더 좋다.
df.pivot_table(index="음주여부", values="가입자일련번호", aggfunc="count")
"음주여부"에 따른 감마지티피의 평균 구하기
pd.pivot_table(df, index="음주여부", values="감마지티피") # 기본적으로 평균값 구함
pd.pivot_table(df, index="음주여부", values="감마지티피", aggfunc='mean')
aggfunc에 여러 값을 한번에 지정할 수도 있다.
pd.pivot_table(df, index="음주여부", values="감마지티피", aggfunc=["mean", "median"]) # 평균값과 중앙값 비교
aggfunc에 describe를 사용해 통계요약값을 한번에 볼 수도 있다.
pd.pivot_table(df, index="음주여부", values="감마지티피", aggfunc="describe")
"성별코드", "음주여부"에 따른 감마지티피 값의 통계요약값을 구함
pd.pivot_table(df, index=["성별코드", "음주여부"], values="감마지티피", aggfunc="describe")
11. 전체 데이터 시각화하기
- 100만개가 넘는 데이터를 시각화를 할 때는 되도록이면 groupby 혹은 pivot_table로 연산을 하고 시각화를 하는 것을 권장
- 100만개가 넘는 데이터를 seaborn과 같은 고급 통계 연산을 하는 그래프를 사용하게 되면 많이 느릴수도 있음
1) 히스토그램
- 판다스의 info 기능을 통해 대부분 수치 데이터로 이루어 진 것을 확인할 수 있음
- 히스토그램을 사용하면 수치데이터를 bin의 갯수만큼 그룹화해서 도수분포표를 만들고 그 결과를 시각화
- 이 데이터는 수치데이터가 많기 때문에 판다스의 hist를 사용해서 히스토그램을 그림
전체 데이터에 대한 히스토그램을 출력하기
h = df.hist(figsize=(12, 12)) # 변수에 할당을 하면 출력되는 결과가 없이 깔끔하게 출력
2) 슬라이싱을 사용해 히스토그램 그리기
- 슬라이싱 기능을 사용해서 데이터를 나누어 그림
- 슬라이싱 사용 시 iloc를 활용하면 인덱스의 순서대로 슬라이싱이 가능
- iloc[행, 열] 순으로 인덱스를 써주면 해당 인덱스만 불러오며, 전체 데이터를 가져오고자 할 때는 [:, :]을 사용
- 슬라이싱을 해주는 대괄호 안의 콜론 앞뒤에 숫자를 써주게 되면 해당 시작인덱스:끝나는인덱스(+1)를 지정할 수 있음
슬라이싱을 사용해 앞에서 12개 컬럼에 대한 데이터로 히스토그램을 그리기 [행, 열]
h = df.iloc[:, :12].hist(figsize=(12, 12))
- 슬라이싱을 사용해 앞에서 12번째부터 23번째까지 (12:24) 컬럼에 대한 데이터로 히스토그램을 그리기
- bins: 막대의 개수 -> 값의 분포를 자세하게 볼 수 있다.
h = df.iloc[:, 12:24].hist(figsize=(12, 12), bins=100)
슬라이싱을 사용해 앞에서 24번째부터 마지막까지 (24:) 컬럼에 대한 데이터로 히스토그램을 그리기
h = df.iloc[:, 24:].hist(figsize=(12, 12), bins=10)
12. 샘플데이터 추출하기
- seaborn의 그래프는 내부에서 수학적 연산이 되기 때문에 데이터가 많으면 속도가 오래 걸린다.
- 따라서 전체 데이터를 사용하면 너무 느리기 때문에 일부만 샘플링해서 사용한다.
- df.sample을 통해 일부 데이터만 샘플데이터를 추출한다.
- random_state를 사용해 샘플링되는 값을 고정할 수 있다. → 실험을 통제하기 위해 고정하기한다.
df_sample = df.sample(1000, random_state=1)
df_sample.shape(1000, 34)
13. 데이터 시각화 도구 Seaborn 사용하기
- https://seaborn.pydata.org/
- seaborn은 https://matplotlib.org/ 을 사용하기 쉽게 만들어졌으며, 간단하게 고급 통계 연산을 할 수 있음
seaborn: statistical data visualization — seaborn 0.11.2 documentation
Seaborn is a Python data visualization library based on matplotlib. It provides a high-level interface for drawing attractive and informative statistical graphics. For a brief introduction to the ideas behind the library, you can read the introductory note
seaborn.pydata.org
14. 범주형(카테고리) 데이터 시각화
- countplot은 범주형 데이터의 수를 더한 값으로 그래프로 표현한다.
- value_counts로 구한 값을 시각화한다고 보면 된다.
1) countplot - 음주여부
value_counts로 음주여부에 따른 그래프 그리기
df["음주여부"].value_counts().plot.bar()
countplot로 음주여부에 따른 그래프 그리기 - 위와 달리 연산없이 그래프를 그릴 수 있다.
sns.countplot(x="음주여부", data=df)
2) hue 옵션 사용하기
- 음주여부에 따른 countplot을 그리고 hue를 사용해 성별코드로 색상을 구분해 그리기 -> hue="성별코드"
- 또, seaborn에서 제공하는 폰트 설정을 사용할 수도 있다.
sns.set(font_scale=1, font="Malgun Gothic") # Window
sns.countplot(data=df, x="음주여부", hue="성별코드")
countplot으로 연령대별 음주여부 보기 -> hue="음주여부"
sns.countplot(data=df, x="연령대코드(5세단위)", hue="음주여부")
3) countplot - 키와 몸무게
- 키와 몸무게는 연속형 데이터라고 볼 수 있다.
- 하지만 이 데이터는 키는 5cm, 체중은 5kg 단위로 되어 있음
- 이렇게 특정 범위로 묶게 되면 연속형 데이터라기보다는 범주형 데이터라고 볼 수 있다.
countplot으로 키를 보기
plt.figure(figsize=(15, 4))
sns.countplot(data=df, x="신장(5Cm단위)")
countplot으로 체중을 보기
plt.figure(figsize=(15, 4))
sns.countplot(data=df, x="체중(5Kg단위)")
- countplot으로 신장(5Cm단위)를 보기
- 성별에 따른 키의 차이를 보기 -> hue="성별코드"

성별에 따른 체중의 차이 보기
plt.figure(figsize=(15, 4))
sns.countplot(data=df, x="체중(5Kg단위)", hue="성별코드")
4) barplot - 수치형 vs 범주형 데이터 시각화
- 콜레스테롤과 연령대코드(5세단위)를 음주여부에 따라 barplot으로 그리기
- 검정막대 : 신뢰구간을 의미
sns.barplot(data=df_sample, x="연령대코드(5세단위)", y="총콜레스테롤", hue="음주여부")
콜레스테롤과 연령대코드(5세단위)를 흡연상태에 따라 barplot으로 그리기
plt.figure(figsize=(15, 4))
sns.barplot(data=df_sample, x="연령대코드(5세단위)", y="총콜레스테롤", hue="흡연상태")
- 트리글리세라이드(중성지방) 에 따른 연령대코드(5세단위)를 음주여부에 따라 barplot으로 그리기
- ci : 신뢰구간 표시 (기본값 -> 95), 신뢰구간 표시하지 않으려면 ci=None
sns.barplot(data=df, x="연령대코드(5세단위)", y="트리글리세라이드", hue="음주여부", ci=None)
연령대코드와 체중(5Kg 단위)을 성별에 따라 barplot으로 그리기
sns.barplot(data=df, x="연령대코드(5세단위)", y="체중(5Kg단위)", hue="성별코드", ci=None)
연령대코드와 체중(5Kg 단위)을 음주여부에 따라 barplot으로 그리기
sns.barplot(data=df, x="연령대코드(5세단위)", y="체중(5Kg단위)", hue="음주여부", ci="sd") # 표준편차 그리기
5) lineplot and pointplot
- 연령대코드(5세단위)에 따른 체중(5Kg 단위)을 성별코드에 따라 lineplot으로 그리기
- ci="sd" -> 표준편차가 그림자로 나타남
plt.figure(figsize=(15, 4))
sns.lineplot(data=df_sample, x="연령대코드(5세단위)", y="체중(5Kg단위)", hue="성별코드", ci="sd")
연령대코드(5세단위)에 따른 신장(5Cm 단위)을 성별코드에 따라 lineplot으로 그리기
plt.figure(figsize=(15, 4))
sns.lineplot(data=df_sample, x="연령대코드(5세단위)", y="신장(5Cm단위)", hue="성별코드", ci="sd")
- 연령대코드(5세단위)에 따른 신장(5Cm단위)을 음주여부에 따라 pointplot과 barplot으로 그리기
- pointplot과 barplot 두 개를 겹쳐서 그릴 수도 있다.
plt.figure(figsize=(15, 4))
sns.barplot(data=df_sample, x="연령대코드(5세단위)", y="신장(5Cm단위)", hue="음주여부", ci="sd")
sns.pointplot(data=df_sample, x="연령대코드(5세단위)", y="신장(5Cm단위)", hue="음주여부", ci="sd")
연령대코드(5세단위)에 따른 신장(5Cm단위)을 성별코드에 따라 pointplot으로 그리기
plt.figure(figsize=(15, 4))
sns.pointplot(data=df_sample, x="연령대코드(5세단위)", y="신장(5Cm단위)", hue="성별코드", ci="sd")
연령대코드(5세단위)에 따른 혈색소를 음주여부에 따라 lineplot으로 그리기
plt.figure(figsize=(15, 4))
sns.lineplot(data=df, x="연령대코드(5세단위)", y="혈색소", hue="음주여부", ci=None)
6) boxplot
- 가공하지 않은 자료 그대로를 이용하여 그린 것이 아니라, 자료로부터 얻어낸 통계량인 5가지 요약 수치로 그린다.
- 5가지 요약 수치 : 기술통계학에서 자료의 정보를 알려주는 아래의 다섯 가지 수치를 의미한다.
- 위 그래프보다 값을 더 자세하게 표현한다.
- 최솟값
- 제 1사분위수
- 제 2사분위수(), 즉 중앙값
- 제 3사분위수()
- 최댓값
- Box plot 이해하기
- boxplot으로 신장(5Cm단위) 에 따른 체중(5Kg 단위)을 그리며, 성별코드에 따라 다른 색상으로 표현되게 한다.
- 다이아몬드 : 이상치를 의미한다.
plt.figure(figsize=(15, 4))
sns.boxplot(data=df, x="신장(5Cm단위)", y="체중(5Kg단위)", hue="성별코드")
7) violinplot
violinplot 신장(5Cm단위)에 따른 체중(5Kg단위)을 그리며, 음주여부에 따라 다른 색상으로 표현하게 된다.
plt.figure(figsize=(15, 4))
sns.violinplot(data=df_sample, x="신장(5Cm단위)", y="체중(5Kg단위)", hue="음주여부")
violinplot의 split 기능을 사용하기 -> 두 개의 값을 하나의 그래프에 붙여서 표현한다.
plt.figure(figsize=(15, 4))
sns.violinplot(data=df_sample, x="신장(5Cm단위)", y="체중(5Kg단위)", hue="음주여부", split=True)
violinplot 연령대코드(5세단위)에 따른 혈색소를 그리며, 음주여부에 따라 다른 색상으로 표현하게 된다.
plt.figure(figsize=(15, 4))
sns.violinplot(data=df_sample, x="연령대코드(5세단위)", y="혈색소", hue="음주여부", split=True)
8) swarm plot
: 범주형 데이터를 산점도로 시각화하고자 할 때 사용한다.
- swarmplot으로 신장(5Cm단위)에 따른 체중(5Kg단위)을 그리며, 음주여부에 따라 다른 색상으로 표현되게 한다.
- 점을 하나씩 찍기 때문에 오래 걸리는 코드는 전체로 그려보기 전에 일부만 가져와 그리기
plt.figure(figsize=(15, 4))
sns.swarmplot(data=df_sample, x="신장(5Cm단위)", y="체중(5Kg단위)", hue="음주여부")
# sns.violinplot(data=df_sample, x="신장(5Cm단위)", y="체중(5Kg단위)") # violinplot과 같이 그릴 수 있음
swarmplot으로 연령대코드(5세단위)에 따른 혈색소를 그리며, 음주여부에 따라 다른 색상으로 표현되게 한다.
plt.figure(figsize=(15, 4))
sns.swarmplot(data=df_sample, x="연령대코드(5세단위)", y="혈색소", hue="음주여부")
- lmplot으로 그리기 - 회귀선을 바탕으로 x축과 y축의 상관관계를 알아본다.
- 장점 : 여러 개의 값을 subplot으로 표현 가능하다.
- col : hue값에 따라서 column을 다르게 표현할 수 있다.
sns.lmplot(data=df_sample, x="연령대코드(5세단위)", y="혈색소", hue="음주여부", col="성별코드")
15. 수치형 데이터 시각화
1) scatterplot - 산점도
- 수치형 vs 수치형 데이터의 상관 관계를 볼 때 주로 사용
- 점의 크기를 데이터의 수치에 따라 다르게 볼 수 있음
- scatterplot으로 "(혈청지오티)AST", "(혈청지오티)ALT" 을 그리고 음주여부에 따라 다른 색상으로 표현한다.
- x축, y축 모두 수치형 데이터이며 체중에 따라서 원의 크기를 다르게 표현할 수 있다. -> size="체중"
plt.figure(figsize=(8, 7))
sns.scatterplot(data=df_sample, x="(혈청지오티)AST", y="(혈청지오티)ALT", hue="음주여부", size="체중(5Kg단위)")
2) Implot - 상관관계를 보기
- Implot으로 신장(5Cm단위)에 따른 체중(5Kg단위)을 그리며, 음주여부에 따라 다른 색상으로 표현한다.
- 장점 : 여러 카테고리의 값에 따라서 서브플롯을 그려볼 수 있음
sns.lmplot(data=df_sample, x="신장(5Cm단위)", y="체중(5Kg단위)", hue="음주여부")
Implot의 col기능을 통해 음주여부에 따라 서브플롯을 그린다.
sns.lmplot(data=df_sample, x="신장(5Cm단위)", y="체중(5Kg단위)", hue="성별코드", col="음주여부")
lmplot으로 수축기, 이완기혈압을 그리고 음주여부에 따라 다른 색상으로 표현한다.
sns.lmplot(data=df_sample, x="수축기혈압", y="이완기혈압", hue="음주여부")
- lmplot으로 "(혈청지오티)AST", "(혈청지오티)ALT" 을 그리고 음주여부에 따라 다른 색상으로 표현한다.
- robust : 이상치의 값이 영향을 적게 받게 그려줌 -> robust=True
sns.lmplot(data=df_sample, x="(혈청지오티)AST", y="(혈청지오티)ALT", hue="음주여부", robust=True)
3) 이상치 다루기
- 이상치가 있으면 데이터가 자세히 보이지 않거나 회귀선이 달라지기도 한다.
- 시각화를 통해 찾은 이상치를 제거하려고 보거나 이상치만 따로 모아 보도록 함
"(혈청지오티)AST"와 "(혈청지오티)ALT"가 400 이하인 값만 데이터프레임 형태로 추출해서 df_ASLT 라는 변수에 담기
df_ASLT = df_sample[(df_sample["(혈청지오티)AST"] < 400) & (df_sample["(혈청지오티)ALT"] < 400)]
df_ASLT
이상치를 제거한 "(혈청지오티)AST"와 "(혈청지오티)ALT"를 lmplot으로 그리며 음주여부에 따라 다른 색으로 표현한다.
-> hue="음주여부", ci=None
sns.lmplot(data=df_ASLT, x="(혈청지오티)AST", y="(혈청지오티)ALT", hue="음주여부", ci=None)
- "(혈청지오티)AST"와 "(혈청지오티)ALT"가 400 이상인 값만 데이터프레임 형태로 추출해서 df_ASLT_high라는 변수에 담는다.
- df_sample에는 조건에 맞는 데이터가 없기 때문에 df로 사용
df_ASLT_high = df[(df["(혈청지오티)AST"] > 400) | (df["(혈청지오티)ALT"] > 400)]
df_ASLT_high
위에서 구한 df_ASLT_high 데이터 프레임에 담겨진 혈청지오티(AST)가 높은 데이터만 따로 본다.
sns.lmplot(data=df_ASLT_high, x="(혈청지오티)AST", y="(혈청지오티)ALT", hue="음주여부", ci=None)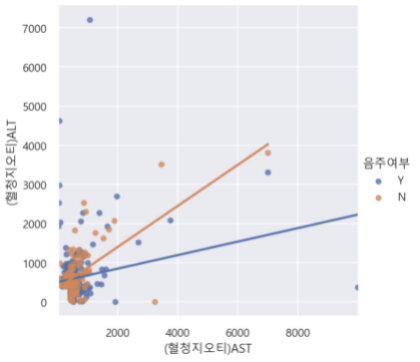
- 위에서 구한 df_ASLT_high 데이터 프레임의 혈청지오티(AST)rk 8000 이상인 데이터만 따로 df_ASLT_high_8000 변수에 담는다.
- df_ASLT_high_8000 데이터 프레임 인덱스 10부터 26까지 추출 -> iloc[:, 10:27]
df_ASLT_high_8000 = df_ASLT_high[df_ASLT_high["(혈청지오티)AST"] > 8000]
df_ASLT_high_8000.iloc[:, 10:27]

4) distplot
우선, 수치형 데이터로 된 컬럼을 찾기 위해 컬럼명만 따로 출력한다.
df.columns
결측치 없는(notnull() 함수) "총콜레스테롤" 데이터를 추출하여 "총콜레스테롤" column만 출력하여 df_chol 라는 변수에 저장한다.
df_chol = df.loc[df["총콜레스테롤"].notnull(), "총콜레스테롤"]
df_chol.head()
df_chol 데이터프레임을 describe() 함수를 이용하여 요약한다.
df_chol.describe()
- "총콜레스테롤"에 따른 distplot을 그린다.
- bins : 막대기 개수
sns.distplot(df_chol)
- 음주여부가 Y인 값에 대한 "총콜레스테롤" 을 distplot으로 그린다.
- displot을 사용할 때는 series 형태를 넣어야 한다. (dataframe 형태 X)
-> 행 : 결측치 없고 & 음주여부 Y인 데이터, 열 : 총콜레스테롤
sns.distplot(df.loc[
df["총콜레스테롤"].notnull() & (df["음주여부"] == "Y"), # 행
"총콜레스테롤"]) # 열
음주여부가 N인 값에 대한 "총콜레스테롤" 을 distplot으로 그리기
sns.distplot(df.loc[
df["총콜레스테롤"].notnull() & (df["음주여부"] == "N"), # 행
"총콜레스테롤"]) # 열
- 음주여부 값에 대한 "총콜레스테롤"을 distplot으로 그리며, 하나의 그래프에 표시되도록 함 (히스토그램 표시 안 함)
- 하나의 그래프에 표시되도록 하려면 kdeplot으로 그릴 수 있다.
plt.axvline(df_sample["총콜레스테롤"].mean(), linestyle=":") # 그래프 가운데 평균값이 그려짐
plt.axvline(df_sample["총콜레스테롤"].median(), linestyle="--") # 중앙값 표시
sns.kdeplot(df_sample.loc[
df_sample["총콜레스테롤"].notnull() & (df["음주여부"] == "Y"), # 행
"총콜레스테롤"], label="음주 중") # 열
sns.kdeplot(df_sample.loc[
df_sample["총콜레스테롤"].notnull() & (df["음주여부"] == "N"), # 행
"총콜레스테롤"], label="음주 안 함") # 열
plt.legend() # 라벨 표시
감마지티피 값에 따라 음주여부 시각화하기
s_1 = df_sample.loc[df_sample["음주여부"] == "Y", "감마지티피"]
s_0 = df_sample.loc[df_sample["음주여부"] == "N", "감마지티피"]
sns.kdeplot(s_1, label="음주 중")
sns.kdeplot(s_0, label="음주 안 함")
plt.legend()
16. 상관분석
상관계수에 사용할 컬럼을 columns 변수에 담는다.
columns = ['연령대코드(5세단위)', '체중(5Kg단위)', '신장(5Cm단위)', '허리둘레', '시력(좌)', '시력(우)', '청력(좌)', '청력(우)',
'수축기혈압', '이완기혈압', '식전혈당(공복혈당)', '총콜레스테롤', '트리글리세라이드', 'HDL콜레스테롤', 'LDL콜레스테롤',
'혈색소', '요단백', '혈청크레아티닌', '(혈청지오티)AST', '(혈청지오티)ALT', '감마지티피', '흡연상태', '음주여부']
1) 상관계수 구하기
샘플컬럼만 가져와서 df_small이라는 데이터프레임에 담은 뒤 상관계수를 구한다. -> corr() 함수 이용
df_small = df_sample[columns]
df_corr = df_small.corr() # default : 피어슨 상관계수
df_corr # 대각선은 모두 1 (자기 자신이기 때문에)
출처: https://www.boostcourse.org/ds112/joinLectures/28143?isDesc=false
파이썬으로 시작하는 데이터 사이언스
부스트코스 무료 강의
www.boostcourse.org
'Data analysis > 실습 & 프로젝트' 카테고리의 다른 글
| [파이썬 데이터 분석 실무 테크닉 100] 1-2. 대리점 데이터를 가공하는 테크닉 10 (0) | 2022.05.28 |
|---|---|
| [파이썬 데이터 분석 실무 테크닉 100] 1-1. 웹에서 주문 수를 분석하는 테크닉 10 (0) | 2022.05.28 |
| 통계청 출생아수 데이터 분석하기 (0) | 2022.02.03 |
| K-beauty 온라인 판매분석 (0) | 2022.01.30 |
| 서울 종합병원 분포 확인하기 (0) | 2022.01.26 |



